Introducción¶
En este ejemplo vamos a construir nuestra propria red trófica de un sistema de coco en Sri Lanka. Para guardar las cosas sencillas no vamos a incluir el cultivo. De todo modo, no tengo modelo de cultivo para árboles de coco.
Aquí está un imagen general de los insectos involucrados (3 en total). Tenemos solamente una plaga, y dos parasitoides se atacan a varias etapas de su ciclo de vida.
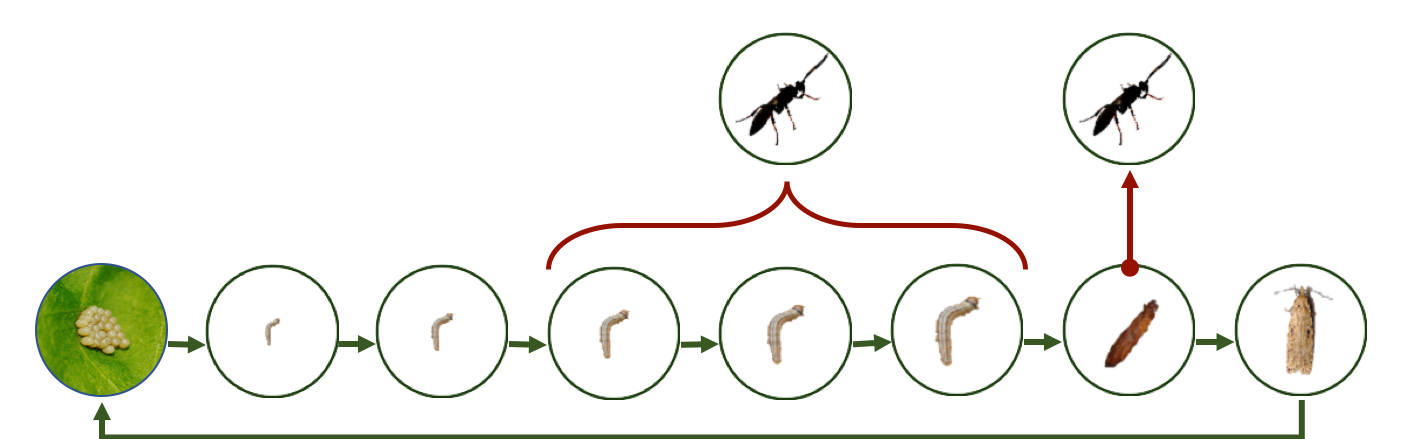
Especificar insectos¶
Primero especificaremos nuestros insectos presentes, una oruga y dos parasitoides (ver Insectos para la lista completa de clases disponibles).
from tikon.rae.orgs.insectos import MetamCompleta, Parasitoide
from tikon.rae.red_ae import RedAE
# Mariposas tienen metamórfosis completa
Oarenosella = MetamCompleta('O. arenosella', njuvenil=5)
# 2 tipos de parasitoides
Paras_larvas = Parasitoide('Parasitoide larvas', pupa=True)
Paras_pupa = Parasitoide('Parasitoide pupa')
# Juntamos todo en una red
with RedAE([Oarenosella, Paras_larvas, Paras_pupa]) as red:
# El parasitoide de larvas parasita las fases 3, 4, y 5 de O. arenosela y emergen después de la quinta
Paras_larvas.parasita(Oarenosella, ['juvenil_3', 'juvenil_4', 'juvenil_5'], etp_emerg='juvenil_5')
# El parasitoide de pupas parasita y emerge de la pupa
Paras_pupa.parasita(Oarenosella, 'pupa', etp_emerg='pupa')
A prioris¶
Un a priori es una distribución probable del valor de un parámetro que especificas antes de correr una calibración. Ayuda el algoritmo de calibración a encontrar el mejor valor del parámetro.
Aquí vamos a cargar las distribuciones a prioris para los parámetros de la red. Los tienes que especificar manualmente para cada experimento.
Advertencia
Es muy importante establecer buenos a prioris para calibraciones. Las limitaciones de algoritmos existentes de calibración de estos tipos de modelos hacen primordial la especificación de a prioris razonables y bastante precisos para todos los parámetros del modelo.
Nota para estudiantes: Mejorar estos algoritmos podría ser buena idea de tesis. :)
En este caso ya especificamos nuestros a prioris en un documento separado así que los vamos a cargar directamente.
from tikon.ejemplos.opisina_arenosella.a_prioris import a_prioris
red.espec_aprioris(a_prioris)
El experimento¶
Aquí se conecta la red con observaciones de campo a través de un experimento (Exper).
from tikon.central import Parcela
from tikon.ejemplos.datos import obt_datos, obt_ref
from tikon.exper.exper import Exper
from tikon.rae.red_ae.obs import ObsPobs
# Datos de observaciones
datos = obt_datos('Perera et al 1988/Oarenosella_A.csv')
# También se puede visualisar la referencia para los datos
print(obt_ref('Perera et al 1988/Oarenosella_A.csv'))
# Se trata de observaciones de poblaciones (y no de otro variable, como depredación).
pobs = ObsPobs.de_csv(
datos,
col_tiempo='Día',
corresp={
'Estado 1': Oarenosella['juvenil_1'],
'Estado 2': Oarenosella['juvenil_2'],
'Estado 3': Oarenosella['juvenil_3'],
'Estado 4': Oarenosella['juvenil_4'],
'Estado 5': Oarenosella['juvenil_5'],
'Pupa': Oarenosella['pupa'],
'Para_larva_abs': Paras_larvas['juvenil'],
'Para_pupa_abs': Paras_pupa['juvenil']
},
factor=655757.1429 / 500 # para convertir a individuos por ha
)
exper_A = Exper('Sitio A', Parcela('Sitio A', geom=GeomParcela((7.297, 79.865))))
exper_A.datos.agregar_obs(pobs)
Calibración¶
Ahora vamos a calibrar nuestro modelo. Primero creamos un Simulador para poder correr
simulaciones y calibraciones. En nuestro ejemplo sencillo el simulador solamente tiene un módulo (la red), pero
se podrían incluir clima, manejo, o cultivos también.
from tikon.central. import Modelo
modelo = Modelo(red)
modelo.calibrar('Sitio A', exper=exper_A)
Validación¶
En este ejemplo vamos a hacer trampa y validar con los mismos datos de calibración.
Primero hacemos una simulación normal, y despues vamos a validar() los
resultados. También los podremos graficar().
Las observaciones especificadas arriba quedaron vinculadas en los resultados y por eso se tomarán en cuenta en la validación y en los gráficos.
res = simul.simular(exper=exper_A)
from pprint import pprint
pprint(res.validar())
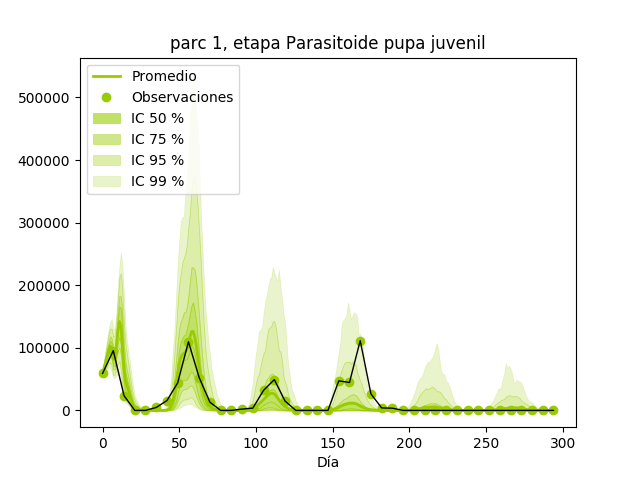
res.graficar('gráficos Sitio A')
Tiko’n generará un gráfico para cada insecto de la red, con su población predicha, los márgenes de incertidumbre y las observaciones, si hay.
Guardar y cargar¶
Vamos a guardar los resultados de la calibración para ahorar tiempo en el futuro. Tiko’n calibra automáticamente las poblaciones iniciales para etapas cuyas poblaciones no se observaron en el experimento, así que guardaremos la calibración del experimento también.
simul.guardar_calib('calibs Sitio A')
exper_A.guardar_calib('calibs Sitio A')
Se pueden después cargar las calibraciones para más trabajo. Igualmente se pueden compartir entre usuarias de Tiko’n. Por ejemplo, en otra sesión de Python:
red.cargar_calib('calibs Sitio A')
exper_A.cargar_calib('calibs Sitio A')
red.simular(exper=exper_A)
Igualmente puedes guardar tu calibración al directorio de Tiko’n. Será después disponible para todas los usuarios de tu instalación de Tiko’n. Si quieres, también lo puedes compartir en GitHub con el resto de la comunidad de Tiko’n.
from tikon.ejemplos.calibs import guardar_calib
guardar_calib(
[red, exper_A],
'Opisina arenosella, Perera et al. 1988',
autor='Yo :)'
correo='julien.malard@mail.mcgill.ca',
detalles='Calibración con Sitio A'
)
Después se podrá acceder con:
from tikon.ejemplos.calibs import obt_calib, obt_ref
dir_ = 'Opisina arenosella, Perera et al. 1988'
red.cargar_calib(obt_calib(dir_))
exper_A.cargar_calib(obt_calib(dir_))
# Visualizar la información de la calibración
print(obt_ref(dir_))